Durante los últimos 50 años se han determinado las formas tridimensionales o conformaciones de más de mil proteínas. Una conformaciónes un ordenamiento espacial de átomos que depende de la rotación de uno o varios enlaces. La conformación de una molécula, como la de una proteína, puede cambiar sin que los enlaces covalentes se rompan, mientras que las diversas configuraciones de una molécula sólo se pueden cambiar si se rompen y vuelven a unir enlaces covalentes. (Recuérdese que las formas L y D de los aminoácidos representan configuraciones diferentes).
. Las proteínas fibrosas son una clase particular de proteínas estructurales que proporcionan soporte mecánico a las células u organismos. En el caso típico, las proteínas fibrosas se ensamblan en grandes cables o hebras. Como ejemplos de proteínas fibrosas están la a-queratina, el componente principal de cabello y uñas, y la colágena, el componente proteínico principal de tendones, piel, huesos y dientes. Entre otros ejemplos de proteínas estructurales están los componentes proteínicos de virus, bacteriófagos, esporas y polen.
Hay cuatro niveles de estructura de las proteínas
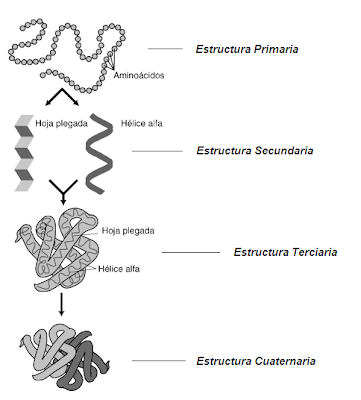
Las moléculas individuales de proteína se pueden describir mediante hasta cuatro niveles de estructura .La estructura primaria describe la secuencia lineal de residuos de aminoácidos en una proteína. Recuérdese que las secuencias de aminoácidos siempre se escriben desde el amino terminal (N-terminal) hasta el carboxilo terminal C- (C-terminal). La estructura tridimensional de una proteína se describe con tres niveles adicionales: estructura secundaria, estructura terciaria y estructura cuaternaria. Las fuerzas que mantienen, o estabilizan, estos tres niveles son no covalentes, de manera primordial. La estructura secundariase refiere a las regularidades en las conformaciones locales mantenidas por puentes de hidrógeno entre los hidrógenos de amida y los oxígenos de carbonilo en la columna vertebral del péptido. Las estructuras secundarias principales son las hélices a y las hebras b (incluyendo las láminas a). Se acostumbra representar las regiones helicoidales a con dibujos que muestran las estructuras de proteínas plegadas; las hebras b se representan con flechas anchas que apuntan desde la dirección N-terminal hacia la C-terminal. La estructura terciaria describe la cadena polipeptídica totalmente plegada y compactada. Muchos polipéptidos plegados consisten en varias unidades distintas unidas por un tramo corto de residuos de aminoácidos, como se ve en la figura 4.1c; a dichas unidades se les conoce como dominios. Las estructuras terciarias se estabilizan por las interacciones de cadenas laterales de aminoácidos en regiones no vecinas de la cadena polipeptídica. La formación de la estructura terciaria acerca partes lejanas de las estructuras primaria y secundaria. Algunas proteínas poseen estructura cuaternaria, que implica la asociación de dos o más cadenas polipeptídica en una multisubunidad, o proteína oligomérica u oligómera. Las cadenas polipeptídicas de una proteína oligómera pueden ser idénticas o distintas.
Métodos para determinar la estructura de las proteínas
La técnica acostumbrada para determinar la conformación tridimensional de una proteína es la cristalografía con rayos X. En esta técnica se apunta un haz de rayos X colimados, o paralelos, a un cristal de moléculas de proteína. Los electrones en el cristal difractan los rayos X, que se registran entonces en una película, o mediante un detector electrónico . El análisis matemático de la figura de difracción produce una imagen de las nubes de electrones que rodean a los átomos en el cristal. Este mapa de densidad electrónica revela la forma general de la molécula y las posiciones de cada uno de los átomos en el espacio tridimensional. Al combinar esos datos con los principios del enlazamiento químico es posible deducir el lugar de todos los enlaces en una molécula y en consecuencia su estructura general. La técnica de cristalografía con rayos X se ha desarrollado hasta el punto en que es posible determinar la estructura de una proteína sin tener un conocimiento preciso de la secuencia de aminoácidos.
Otra técnica para analizar la estructura macromolecular de las proteínas es la espectroscopia de resonancia magnética nuclear (RMN) (o NMR, de nuclear magnetic resonance). Este método permite estudiar las proteínas en solución, por lo que no requiere la tediosa preparación de cristales. En la espectroscopia de RMN se coloca una muestra de la proteína en un campo magnético. Ciertos núcleos atómicos absorben radiación electromagnética cuando el campo magnético aplicado se varía.
Conformación del grupo peptídico
Los enlaces de péptido, que unen a los aminoácidos en una cadena polipeptídica. Los dos átomos que intervienen en el enlace peptídico, junto con sus cuatro sustituyentes (el átomo de oxígeno carbonílico, el átomo de hidrógeno de amida y los dos átomos adyacentes de carbono a) constituyen el grupo peptídico. El grupo peptídico es polar.
Debido a la naturaleza del doble enlace en el enlace peptídico, la conformación del grupo peptídico se restringe a una de dos conformaciones posibles, que puede ser trans o cis . En la conformación trans, los dos carbonos a de residuos adyacentes de aminoácido están en lados opuestos del enlace peptídico y en las esquinas opuestas del rectángulo que forma el grupo peptídico plano. En la conformación cis, los dos carbonos a están en el mismo lado del enlace peptídico y están más cerca entre sí. Las conformaciones cis y trans se producen durante la síntesis de la proteína, cuando el enlace peptídico se forma uniendo dos aminoácidos a la cadena polipeptídica. La conformación cis es menos favorable que la conformación trans, que es extendida debido a impedimentos estéricos entre las cadenas laterales unidas a los dos átomos de carbono a.
- Estructura Secundaria
 En teoría, una hélice a puede ser una rosca izquierda o derecha. Las hélices a que se encuentran en las proteínas casi siempre son derechas. La mayor parte de las hélices a está ligeramente distorsionada en las proteínas, pero en general tienen entre 3.5 y 3.7 residuos por vuelta.
En teoría, una hélice a puede ser una rosca izquierda o derecha. Las hélices a que se encuentran en las proteínas casi siempre son derechas. La mayor parte de las hélices a está ligeramente distorsionada en las proteínas, pero en general tienen entre 3.5 y 3.7 residuos por vuelta.
Dentro de una hélice a, cada residuo con oxígeno carbonílico (residuo n) de la columna vertebral del polipéptido está unido mediante puente de hidrógeno con el hidrógeno de la amida en la columna vertebral del cuarto residuo siguiendo la dirección hacia el C-terminal (residuo n 4). (Los tres grupos amino en un extremo de la hélice y los tres grupos carbonilo en el otro extremo carecen de receptores de puente de hidrógeno dentro de la hélice). Cada puente de hidrógeno cierra un asa que contiene 13 átomos —el oxígeno carbonílico, 11 átomos de columna vertebral y el hidrógeno de amida.
Muchas hélices a muestran aminoácidos hidrofílicos en una cara del cilindro de la hélice y aminoácidos hidrofóbicos en la cara opuesta. Es fácil comprobar la naturaleza anfipática de la hélice cuando se traza la secuencia de aminoácidos como una espiral, llamada rueda helicoidal.
Hebras beta y láminas beta
La otra estructura secundaria común se llama estructura b, una clase que incluye a hebras b y láminas b. Las hebras B son partes de la cadena polipeptídica que se encuentran casi totalmente extendidas.
Cuando se ordenan varias hebras b lado a lado forman láminas B.
Asas y giros
En una hélice a o en una hebra b, los residuos consecutivos tienen una conformación similar que se repite en toda la estructura. También, las proteínas presentan tramos de estructura tridimensional no repetitiva. La mayor parte de esas regiones de estructura secundaria se puede caracterizar como rizos (o asas) y giros (o vueltas) porque causan cambios de dirección en la columna vertebral del polipéptido.
Las asas y los giros unen a hélices a y hebras b y permiten que la cadena de polipéptido se doble sobre sí misma para producir la forma tridimensional compacta que se ve en la estructura nativa.
Las asas contienen con frecuencia residuos hidrofílicos y se suelen encontrar en las superficies de las proteínas, donde están expuestas al solvente y forman puentes de hidrógeno con el agua.
Las asas que sólo contienen pocos (hasta cinco) residuos se llaman giros si causan un cambio abrupto en la dirección de una cadena de polipéptidos. Los tipos más comunes de giros bruscos se llaman giros inversos o giros B porque con frecuencia conectan hebras b antiparalelas diferentes.
La estructura terciaria se debe al plegamiento de un polipéptido (que puede tener ya algunas regiones de hélice a y de estructura b) para formar una estructura tridimensional empacada en forma apretada. Una propiedad importante de la estructura terciaria es que los residuos de aminoácidos alejados en la estructura primaria se acercan entre sí y permiten interacciones entre sus cadenas laterales. La estructura terciaria se halla estabilizada en especial por interacciones no covalentes.
También los puentes disulfuro, aunque son covalentes, son elementos de la estructura terciaria (no son parte de la estructura primaria porque sólo se forman después que la proteína se dobla).
A. Estructuras supersecundarias
Las estructuras supersecundarias, o motivos, son combinaciones reconocibles de hélices a, hebras b y giros que aparecen en diversas proteínas. A veces los motivos se relacionan con determinada función, aunque los motivos de estructura similar pueden tener funciones distintas en proteínas diferentes.
B. Dominios
Hay muchas proteínas que están formadas por varias unidades compactas, discretas, plegadas en forma independiente llamadas dominios. Los dominios pueden consistir en combinaciones de motivos. El tamaño de un dominio varía desde unos 25 a 30 residuos de aminoácidos hasta más de 300.
Los dominios están unidos por asas, pero también se unen entre sí mediante interacciones débiles formadas por las cadenas laterales de aminoácidos en la superficie de cada dominio. Los dominios de proteína pueden seguirse agrupando por la presencia de dobleces característicos. Un doblezo plieguees una combinación de estructuras secundarias que forman el núcleo de un dominio.
Hebras beta y láminas beta
La otra estructura secundaria común se llama estructura b, una clase que incluye a hebras b y láminas b. Las hebras B son partes de la cadena polipeptídica que se encuentran casi totalmente extendidas.
Cuando se ordenan varias hebras b lado a lado forman láminas B.
Las hebras b en una lámina pueden ser paralelas (con la misma dirección de N a C-terminal) o antiparalelas (con direcciones opuestas de N a C-terminal). Cuando las hebras b son antiparalelas, los puentes de hidrógeno son casi perpendiculares a las cadenas extendidas del polipéptido. Nótese que en la lámina b antiparalela los átomos de oxígeno carbonílico y los de hidrógeno de amida de un residuo forman puentes de hidrógeno con el hidrógeno de amida y el oxígeno carbonílico de un solo residuo en la otra hebra.
Las láminas paralelas son menos estables que las antiparalelas, quizá porque los puentes de hidrógeno están distorsionados en el ordenamiento paralelo. A veces, a la lámina b se le llama lámina B plegada ya que los grupos peptídicos planos se encuentran entre sí formando ángulos como en el plisado de un acordeón.
Asas y giros
En una hélice a o en una hebra b, los residuos consecutivos tienen una conformación similar que se repite en toda la estructura. También, las proteínas presentan tramos de estructura tridimensional no repetitiva. La mayor parte de esas regiones de estructura secundaria se puede caracterizar como rizos (o asas) y giros (o vueltas) porque causan cambios de dirección en la columna vertebral del polipéptido.
Las asas y los giros unen a hélices a y hebras b y permiten que la cadena de polipéptido se doble sobre sí misma para producir la forma tridimensional compacta que se ve en la estructura nativa.
Las asas contienen con frecuencia residuos hidrofílicos y se suelen encontrar en las superficies de las proteínas, donde están expuestas al solvente y forman puentes de hidrógeno con el agua.
Las asas que sólo contienen pocos (hasta cinco) residuos se llaman giros si causan un cambio abrupto en la dirección de una cadena de polipéptidos. Los tipos más comunes de giros bruscos se llaman giros inversos o giros B porque con frecuencia conectan hebras b antiparalelas diferentes.
- Estructura Terciaria de las Proteínas
La estructura terciaria se debe al plegamiento de un polipéptido (que puede tener ya algunas regiones de hélice a y de estructura b) para formar una estructura tridimensional empacada en forma apretada. Una propiedad importante de la estructura terciaria es que los residuos de aminoácidos alejados en la estructura primaria se acercan entre sí y permiten interacciones entre sus cadenas laterales. La estructura terciaria se halla estabilizada en especial por interacciones no covalentes.
También los puentes disulfuro, aunque son covalentes, son elementos de la estructura terciaria (no son parte de la estructura primaria porque sólo se forman después que la proteína se dobla).
Las estructuras supersecundarias, o motivos, son combinaciones reconocibles de hélices a, hebras b y giros que aparecen en diversas proteínas. A veces los motivos se relacionan con determinada función, aunque los motivos de estructura similar pueden tener funciones distintas en proteínas diferentes.
B. Dominios
Hay muchas proteínas que están formadas por varias unidades compactas, discretas, plegadas en forma independiente llamadas dominios. Los dominios pueden consistir en combinaciones de motivos. El tamaño de un dominio varía desde unos 25 a 30 residuos de aminoácidos hasta más de 300.
Los dominios están unidos por asas, pero también se unen entre sí mediante interacciones débiles formadas por las cadenas laterales de aminoácidos en la superficie de cada dominio. Los dominios de proteína pueden seguirse agrupando por la presencia de dobleces característicos. Un doblezo plieguees una combinación de estructuras secundarias que forman el núcleo de un dominio.
C. Estructura y función de los dominios
Es compleja la relación entre estructura y función de un dominio. Con frecuencia, un solo dominio tiene determinada función, como por ejemplo unirse a pequeñas moléculas o catalizar una sola reacción. En las enzimas multifuncionales, cada actividad catalítica puede estar asociada con uno de varios dominios presentes en una sola cadena polipeptídica. Sin embargo, en muchos casos el enlazamiento de moléculas pequeñas y la formación del sitio activo de una enzima se llevan a cabo en la interfase entre dos dominios separados. Estas interfases suelen formar grietas, hendiduras y bolsas a las que se puede llegar en la superficie de la proteína. El grado de contacto entre dominios varía de una proteína a otra.
- Estructura Cuaternaria
Estructura cuaternaria se refiere a la organización y el ordenamiento de subunidades en una proteína con múltiples subunidades. Cada subunidad es una cadena polipeptídica aparte. Una proteína con varias subunidades, o de multisubunidades, se llama oligómero (las proteínas que sólo tienen una cadena polipeptídica son monómeros). Las subunidades integrantes de cada proteína oligomérica tienen siempre una estequiometría definida y el ordenamiento de las subunidades produce una estructura estable. Las subunidades de una proteína de multisubunidades pueden ser idénticas o diferentes.
El hecho de que una gran proporción de proteínas consista en subunidades múltiples se relaciona, probablemente, con varios factores.
1. Los oligómeros suelen ser más estables que sus subunidades disociadas, lo que parece indicar que la estructura cuaternaria prolonga la duración in vivo de una proteína.
2. Los sitios activos de algunas enzimas oligoméricas se forman con residuos procedentes de cadenas de polipéptido adyacentes.
3. Las estructuras tridimensionales de muchas proteínas oligoméricas cambian cuando éstas se unen con ligandos. Tanto las estructuras terciarias de las subunidades como las cuaternarias (es decir, los contactos entre subunidades) se pueden alterar. Algunos cambios son elementos clave en la regulación de la actividad biológica de ciertas proteínas oligoméricas.
4. Proteínas diferentes pueden compartir las mismas subunidades. Como muchas subunidades desempeñan una función definida (por ejemplo, unión con el ligando), la evolución ha favorecido la selección de diferentes combinaciones de subunidades para efectuar funciones relacionadas. Dicha elección es más eficiente que la selección de una proteína monomérica totalmente nueva que duplique parte de la función.
Desnaturalización y renaturalización de las proteínas
Los cambios en el ambiente o los tratamientos químicos pueden alterar la conformación nativa de una proteína con la pérdida concomitante de su actividad biológica. Esa alteración se llama desnaturalización. La cantidad de energía necesaria para causar la desnaturalización es pequeña.
Las proteínas se suelen desnaturalizar por calentamiento. Bajo condiciones adecuadas, un aumento modesto de temperatura causa el desdoblamiento y pérdida de las estructuras secundaria y terciaria. El desdoblado es un proceso cooperativo: la desestabilización de sólo unas pocas interacciones débiles causa una pérdida casi completa de la conformación nativa.
Bajo condiciones fisiológicas, la mayor parte de las proteínas son estables hasta temperaturas de 50 a 60°C.
Las proteínas también se pueden desnaturalizar con dos tipos de sustancias químicas: agentes caotrópicos y detergentes (sección 2.4). Altas concentraciones de agentes caotrópicos, como urea y sales de guanidinio (figura 4.27), desnaturalizan a las proteínas porque permiten que las moléculas de agua solvaten a grupos no polares en su interior.
La desnaturalización completa de las proteínas que contienen puentes de disulfuro requiere romper esos enlaces, además de la alteración de interacciones hidrofóbicas y de los puentes de hidrógeno. El 2-mercaptoetanol u otros reactivos tioles se pueden agregar a un medio desnaturalizante para reducir todos los puentes de disulfuro a grupos sulfhidrilo.
Plegado de proteínas y estabilidad
A medida que el polipéptido recién sintetizado emerge del ribosoma, se pliega en su forma tridimensional característica. Se cree que a medida que una proteína se pliega, el primer par de interacciones inicia otras interacciones que contribuyen al alineamiento de grupos. Este proceso se llama cooperatividad de plegamiento y representa el fenómeno por el que la formación de una parte de una estructura causa la formación de las partes restantes. En su conformación estable final, la proteína nativa es mucho menos sensible a la degradación que una cadena de polipéptido extendida y desdoblada.
El plegado y la estabilización de las proteínas dependen de varias fuerzas no covalentes, como el efecto hidrofóbico, los puentes de hidrógeno, las interacciones de van der Waals y las interacciones entre cargas. Aunque las interacciones no covalentes son débiles en lo individual, en lo colectivo determinan la estabilidad de las conformaciones nativas de las proteínas.
A. El efecto hidrofóbico
estas cadenas son impulsadas a vincularse entre sí, lo cual determina que la cadena polipeptídica se colapse y forme un glóbulo fundido más compacto. Durante el plegado, la disminución de entropía del polipéptido más que se contrarresta por el aumento de la entropía del solvente, cuando se liberan moléculas de agua que estaban unidas a la proteína. (El plegado también altera las jaulas extendidas de moléculas de agua que rodean a los grupos hidrofóbicos). Este aumento general de entropía constituye la principal fuerza impulsora del plegado.
B. Puentes de hidrógeno
Los puentes de hidrógeno contribuyen a la cooperatividad del plegamiento y ayudan a estabilizar las conformaciones nativas de las proteínas. Los primeros puentes de hidrógeno son los que se forman en las hélices a, las láminas b y los giros, y forman regiones definidas de la estructura secundaria.
C. Interacciones de van der Waals e interacciones entre cargas
Los contactos de van der Waals entre cadenas laterales no polares también contribuyen a la estabilidad de las proteínas. Es probable que el efecto acumulado de varias interacciones de van der Waals represente una contribución apreciable a la estabilidad porque las cadenas laterales no polares en el interior de una proteína están empacadas en forma densa.
D. Los chaperones
El doblamiento de proteínas no implica una búsqueda aleatoria en el espacio tridimensional para llegar a la conformación nativa. En lugar de ello, parece que el plegado de proteínas es un proceso cooperativo y secuencial donde la formación de los primeros elementos estructurales ayuda al alineamiento de las características estructurales siguientes.
Los chaperones aumentan la velocidad de plegamiento correcto de algunas proteínas al unir polipéptidos recién sintetizados antes que se doblen por completo. Evitan la formación de intermediarios mal doblados que puedan atrapar al polipéptido en una forma aberrante.
El papel de las proteínas de choque por calor, que hoy se denominan chaperones, es reparar el daño causado por aumentos de temperatura al unirse a proteínas desnaturalizadas y ayudarles a volver a doblarse con rapidez y llegar a su conformación nativa.
La colágena, una proteína fibrosa
La colágena es la principal proteína componente del tejido conectivo de los vertebrados; constituye de 25 a 35% de las proteínas totales en los mamíferos. Las moléculas de colágena tienen formas y funciones notablemente distintas. Por ejemplo, en los tendones forman fibras rígidas, encordadas, de una notable resistencia a la tensión; en la piel, la colágena adquiere la forma de fibras tejidas de forma holgada que permiten la expansión en todas direcciones
La molécula consiste en tres cadenas helicoidales izquierdas enrolladas entre sí para formar una superhélice derecha. Cada hélice izquierda en la colágena presenta 3.0 residuos de aminoácidos por vuelta, y su paso es de 0.94 nm, que implica un ascenso de 0.31 nm por molécula. En consecuencia, una hélice de colágena es más extendida que una hélice a.
Estructuras de la mioglobina y la hemoglobina
Como la mayor parte de las proteínas, la mioglobina (Mb) y la hemoglobina (Hb), una proteína relacionada, desempeñan sus funciones biológicas al unirse en forma selectiva y reversible a otras moléculas —en este caso, al oxígeno molecular (O2). La mioglobina es una proteína monomérica relativamente pequeña que facilita la difusión del oxígeno en los vertebrados. Es la responsable de suministrar oxígeno a los tejidos musculares en los reptiles, aves y mamíferos. La hemoglobina es una proteína tetramérica mayor que transporta el oxígeno en la sangre.
El componente polipeptídico en la mioglobina es miembro de una familia de proteínas llamadas globinas. La estructura terciaria de la mioglobina del esperma de ballena muestra que la proteína consiste en un haz de ocho hélices a.
 El interior de la mioglobina está formado en forma casi exclusiva por residuos hidrofóbicos de aminoácidos, en especial los que son muy hidrofóbicos como valina, leucina, isoleucina, fenilalanina y metionina. La superficie de la proteína contiene residuos tanto hidrofílicos como hidrofóbicos. Como en el caso de la mayor parte de las proteínas, la estructura terciaria de la mioglobina se halla estabilizada por interacciones hidrofóbicas con el interior. El plegado de la cadena de polipéptido está impulsado por la minimización de energía causada por la formación de este interior hidrofóbico.
El interior de la mioglobina está formado en forma casi exclusiva por residuos hidrofóbicos de aminoácidos, en especial los que son muy hidrofóbicos como valina, leucina, isoleucina, fenilalanina y metionina. La superficie de la proteína contiene residuos tanto hidrofílicos como hidrofóbicos. Como en el caso de la mayor parte de las proteínas, la estructura terciaria de la mioglobina se halla estabilizada por interacciones hidrofóbicas con el interior. El plegado de la cadena de polipéptido está impulsado por la minimización de energía causada por la formación de este interior hidrofóbico.Estructuras de las Proteínas
